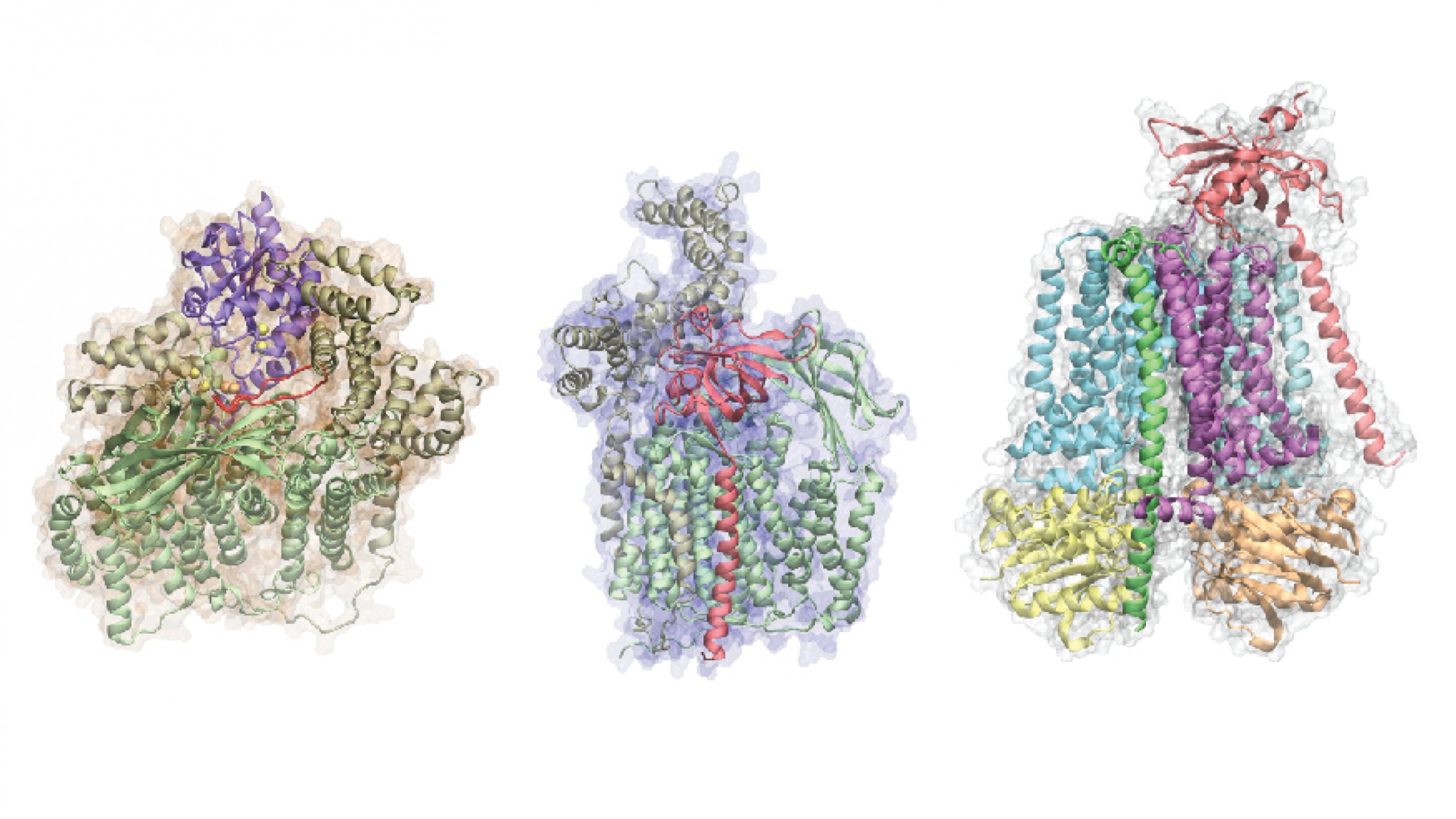
Representación 3D de estructuras complejas de proteínas predichas a partir de secuencias de proteínas por AF2Complex. Crédito: Mu Gao/Tecnología de Georgia
Desde las fibras musculares que nos transmiten hasta las enzimas que copian nuestro ADN, las proteínas son la maquinaria molecular que hace posible la vida.
La función de una proteína depende en gran medida de su estructura tridimensional, e investigadores de todo el mundo han buscado durante mucho tiempo responder a una pregunta aparentemente simple para relacionar la función y la forma: si conociera los componentes básicos de estas máquinas moleculares, ¿puede anticipar cómo? ensamblarlos en su forma funcional?
Esta pregunta no es fácil de responder. Con estructuras complejas que se basan en interacciones físicas complejas, los investigadores han recurrido a modelos de redes neuronales artificiales (marcos matemáticos que transforman patrones complejos en representaciones digitales) para predecir y «ver» cómo se ven las proteínas en una imagen 3D.
En un nuevo artículo publicado en Comunicaciones de la naturalezalos investigadores de Georgia Tech y Oak Ridge National Laboratory se basaron en uno de esos modelos, AlphaFold 2, para predecir no solo la conformación biológicamente activa de proteínas individuales, sino también el apareamiento de proteínas funcionales conocidas como complejos.
Jeffrey Skolnick, profesor de Regentes y presidente de Mary y Maisie Gibson en la Facultad de Ciencias Biológicas y uno de los autores del estudio, dijo que el trabajo podría ayudar a los investigadores a ir más allá de los experimentos prolongados para estudiar la estructura y las interacciones de los complejos de proteínas a escala. , y agregó que tales modelos computacionales podrían significar grandes cosas para el campo.
Si tienen éxito, dijo Skolnik, estos nuevos modelos computacionales «podrían cambiar fundamentalmente la forma en que se estudian los sistemas biomoleculares».

El desarrollo inicial de AF2Complex se llevó a cabo en el Centro de Computación de Asociación para un Entorno de Computación Avanzada (PACE) de Georgia Tech, que se muestra aquí en el centro de datos CUDA. Crédito: Paul Manu/RITMO.
Preparándose para predecir la proteína
Creado por el laboratorio de inteligencia artificial con sede en Londres DeepMind, AlphaFold es un modelo de red neuronal de aprendizaje profundo diseñado para predecir la estructura 3D de una sola proteína al observar su secuencia de aminoácidos. Skolnik y su compañero autor, Mu Gao, investigador sénior de la Facultad de Ciencias Biológicas, compartieron que el programa Alphafold 2 tuvo mucho éxito en las pruebas ciegas que se llevaron a cabo en 14y Réplica de la prueba comunitaria sobre evaluación crítica de técnicas de predicción de estructura de proteínas, o CASP14, una competencia bianual donde investigadores de todo el mundo se reúnen para poner a prueba sus modelos computacionales.
“Para nosotros, lo sorprendente de AlphaFold 2 es que no solo hace excelentes predicciones sobre dominios proteicos individuales (las unidades estructurales o funcionales básicas de la secuencia proteica), sino que también funciona muy bien en secuencias proteicas que constan de múltiples dominios”, Skolnick compartido. Y así, con la capacidad de predecir la estructura de estas proteínas complejas de múltiples dominios, el equipo de investigación se dispuso a determinar si el software podía ir un poco más allá.
Las interacciones físicas entre los diferentes [protein] Zhao explicó que los dominios con la misma secuencia son esencialmente las mismas interacciones que unen diferentes proteínas. Davi Nakajima-Ann, estudiante de cuarto año de la Facultad de Ciencias de la Computación, es reclutada para unirse a los esfuerzos del equipo.
En lugar de comunicar las características de una sola secuencia de proteínas en AlphaFold 2 según su diseño original, los investigadores unieron las características de entrada de múltiples secuencias de proteínas. En combinación con las nuevas métricas para evaluar la fuerza de las interacciones entre las proteínas investigadas, se creó su nuevo programa AF2Complex.

Centro de Supercomputación Summit en el Laboratorio Nacional de Oak Ridge. Crédito: Laboratorio Nacional de Oak Ridge.
Dibujar una nueva área
Para poner a prueba el AF2Complex, los investigadores se asociaron con el Center for High Performance Computing, Partnership for an Advanced Computing Environment (PACE), en Georgia Tech, y encargaron al modelo que predijera las estructuras de los complejos de proteínas nunca antes vistas. El software modificado pudo predecir correctamente la estructura de más del doble de complejos de proteínas que el método tradicional llamado acoplamiento. Si bien AF2Complex solo necesita secuencias de proteínas como entrada, el acoplamiento se basa en el conocimiento previo de las estructuras de proteínas individuales para predecir su estructura combinada en función de motivos complementarios.
«Alentados por estos resultados prometedores, hemos extendido esta idea a un problema mayor, que es predecir las interacciones entre varias proteínas seleccionadas al azar, por ejemplo, en un caso simple, dos proteínas al azar», compartió Skolnick.
Además de predecir la estructura de los complejos de proteínas, AF2Complex se encargó de identificar cuáles de los más de 500 pares de proteínas eran capaces de formar complejos. Utilizando métricas de nuevo diseño, AF2Complex supera los métodos de acoplamiento tradicionales y AlphaFold 2 en la identificación experimental de pares arbitrarios que se sabe que interactúan.
Para probar el AF2Complex en la escala del proteoma, que incluye la biblioteca completa de proteínas de un organismo que se puede expresar, los investigadores recurrieron a Summit Oak Ridge Leadership Computing Facility, el segundo centro de supercomputación más grande del mundo. «Gracias a este recurso, pudimos aplicar AF2Complex a unos 7000 pares de proteínas de la bacteria E. coli», compartió Gao.
Gao dijo que el nuevo modelo del equipo en esta prueba no solo identificó varios pares de proteínas que se sabe que forman complejos, sino que también pudo proporcionar información sobre interacciones «sospechadas pero no observadas experimentalmente».
Profundizar en estas interacciones ha revelado un mecanismo molecular potencial para los complejos de proteínas que son particularmente importantes para la transferencia de energía. Se sabe que estos complejos de proteínas transportan hemos, los metabolitos esenciales que le dan a la sangre su color rojo oscuro. Usando los modelos estructurales predichos del AF2Complex, Jerry M. Gao dijo: «Estos modelos computacionales ahora brindan información sobre los mecanismos moleculares de cómo funciona este sistema biomolecular».
«El aprendizaje profundo está cambiando la forma en que uno estudia un sistema biológico», agregó Skolnik. «Visualizamos métodos como AF2Complex que se convertirán en herramientas poderosas para cualquier biólogo que quiera comprender los mecanismos moleculares de un sistema vivo que incluye interacciones de proteínas».
Mu Gao et al, AF2Complex predice interacciones físicas directas en múltiples proteínas con aprendizaje profundo, Comunicaciones de la naturaleza (2022). DOI: 10.1038 / s41467-022-29394-2
Complejo AF2: github.com/FreshAirTonight/af2complex
La frase: Los investigadores aprovechan el aprendizaje profundo para predecir las interacciones físicas de los complejos de proteínas (18 de abril de 2022) Recuperado el 18 de abril de 2022 de https://phys.org/news/2022-04-leverage-deep-physical-interactions-protein .html
Este documento está sujeto a derechos de autor. Sin perjuicio de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin permiso por escrito. El contenido se proporciona únicamente con fines informativos.